Small RNA是一种小的非编码RNA,长度为18-30个核苷酸,几乎存在于所有的生物体中,包括:miRNA、siRNA和piRNA。Small RNA通过多种多样的作用途径,包括mRNA降解、翻译抑制、异染色质形成以及DNA去除,来调控生物体的生长发育和疾病发生,在人、动植物的转录和转录后调控过程中起着重要的作用。
Small RNA高通量测序分析目前被广泛应用于动植物生长、分化、发育和凋亡等发育调控研究;药物、胁迫等环境适应性研究;病毒、真菌、细菌等免疫互作研究等方面。
小RNA测序分析平台可以进行标准分析和个性化分析,其中标准分析包括:miRNA的鉴定与预测;miRNA表达量分析;miRNA靶基因预测;miRNA靶基因注释分类及富集等。设定参数后点击提交进行分析,分析完成后在基本分析页面下生成标准化结题报告,实现一键式生成。
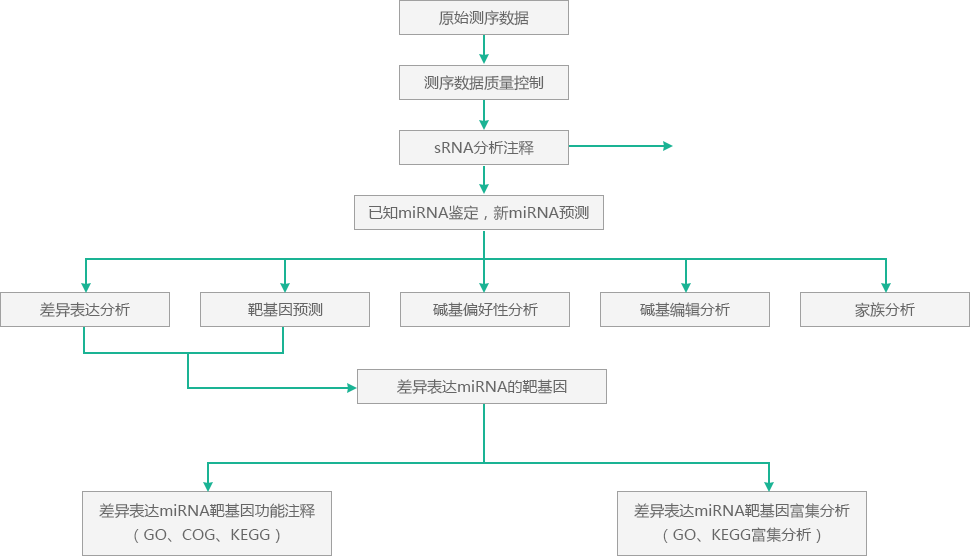
sRNA基本分析以新一代高通量转录组测序(RNA-Seq)数据作为输入,测序得到的原始序列含有接头序列或低质量序列,为了保证信息分析的准确性,需要对原始数据进行质量控制,得到高质量序列(即Clean Reads),原始序列质量控制的标准为:
1.去除低质量Reads(质量值低于30的碱基所占比例超过20%);
2.去除未知碱基N含量大于等于10%的Reads;
3.截除3’端接头和Barcode序列;
4.去除短于18或长于30个核苷酸的序列。
对于得到的Clean Reads,利用Bowtie[1]软件,将其分别与Silva数据库、GtRNAdb数据库、Rfam数据库和Repbase数据库进行序列比对,过滤核糖体RNA(rRNA)、转运RNA(tRNA)、核内小RNA(snRNA)、核仁小RNA(snoRNA)等ncRNA以及重复序列,获得包含miRNA的Unannotated reads。
利用miRDeep2软件[2]将Unannotated reads与参考基因组进行序列比对,获取在参考基因组上的位置信息,即为Mapped Reads。
针对miRNA的生物特征,对于比对到参考基因组的序列,利用miRDeep2软件进行已知及新的miRNA鉴定。miRDeep2软件将18 30nt的核苷酸序列比对到miRBase数据库中特定物种上,鉴定该物种已知的miRNA;过滤获得的未比对到参考基因组,通过碱基数目延伸,进行miRNA结构预测,获得新的miRNA。
miRNA的鉴定和预测完成后,接下来是对各样本中miRNA进行表达量的统计,并用TPM算法[3]对表达量进行归一化处理。TPM归一化处理公式为:

公式中,readcount表示比对到某一miRNA的reads数目;Mapped Reads表示比对到参考基因组上的reads数目。
利用TPM算法对表达量进行归一化处理后,能够消除不同样品间测序量差异的影响,科学、准确的比较各样品间miRNA的差异表达情况。
检测差异表达miRNA时,需要根据实际情况选取合适的差异表达分析软件。DESeq[4]适用于有生物学重复的实验,可以进行样品组间的差异表达分析,获得两个生物学条件之间的差异表达miRNA集;对于没有生物学重复的实验,则使用IDEG6[5]进行差异表达分析,获得两个样品之间的差异表达miRNA。
在差异表达miRNA检测过程中,使用差异倍数(Fold Change,FC)和错误发现率(False Discovery Rate,FDR)作为筛选标准。FC表示两样品(组)间表达量的比值。由于miRNA的差异表达分析是对大量的miRNA表达量进行独立的统计假设检验,会存在假阳性问题,因此在分析过程中,采用了公认的Benjamini Hochberg校正方法对原有假设检验得到的显著性p值(p value)进行校正,并最终采用FDR作为差异表达miRNA筛选的关键指标。
根据已知miRNA和新预测的miRNA与对应物种的基因序列信息,对于植物物种,我们使用TargetFinder软件[6]进行靶基因预测;对于动物物种,用miRanda[7]和RNAhybrid软件[8]进行靶基因预测,对两个软件的预测结果取交集。
靶基因预测完成后,我们会使用BLAST软件将预测到的靶基因与NR[9]、Swiss Prot[10]、GO[11]、COG[12]、KEGG[13]、KOG[14]、Pfam[15]数据库比对,获得靶基因的注释信息。
小RNA在疫霉菌感染大豆中的防御机制解析
本研究对大豆抗病材料 Williams82 和感病材料Harosoy接种大豆疫霉菌,未接种和接种后 8 小时根部进行小 RNA测序,每个样品测序数据量 10-13M。共鉴定到已知miRNA 324 个,属于 109 个miRNA家族。预测到新miRNA 54个,与已有大豆降解组数据比较发现 36 个miRNA具有较高可信度。miR-1507 和 miR-2109 调控 NB-LRR 基因的表达;NB-LRR 和 PPR 基因产生的phasiRNA在侵染后显著上调。这些miRNA和phasiRNA的生物学功能证明在疫霉菌感染大豆中的防御中有重要的调控作用。
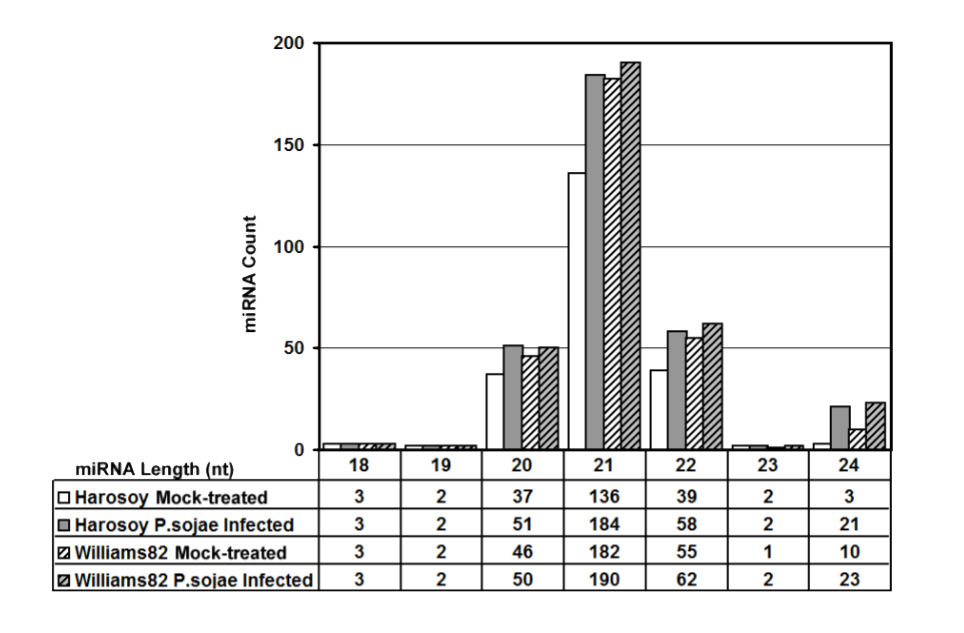
图1.miRNA 长度分布统计
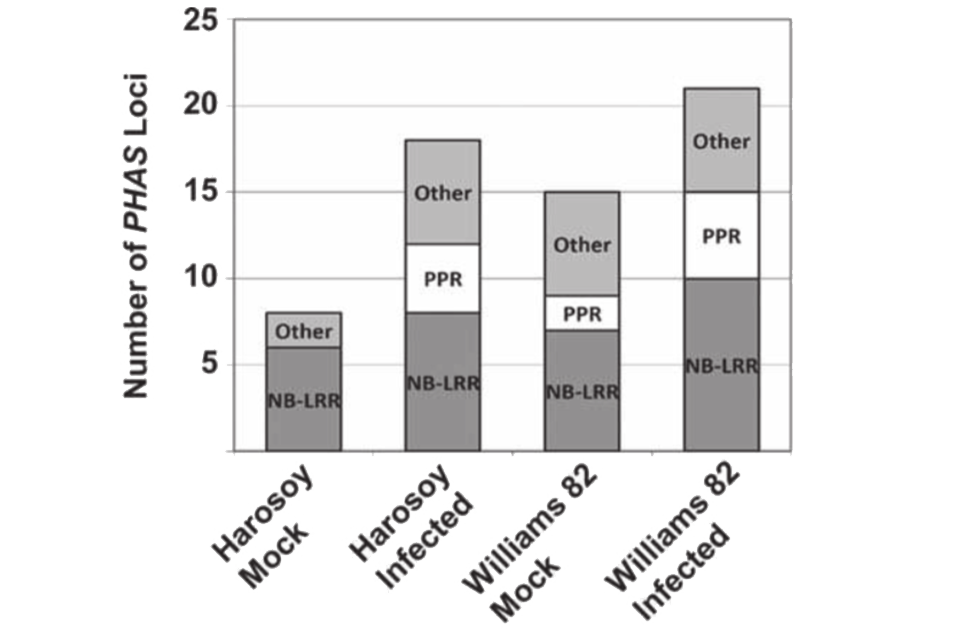
图2.phasiRNA 及其前体基因统计