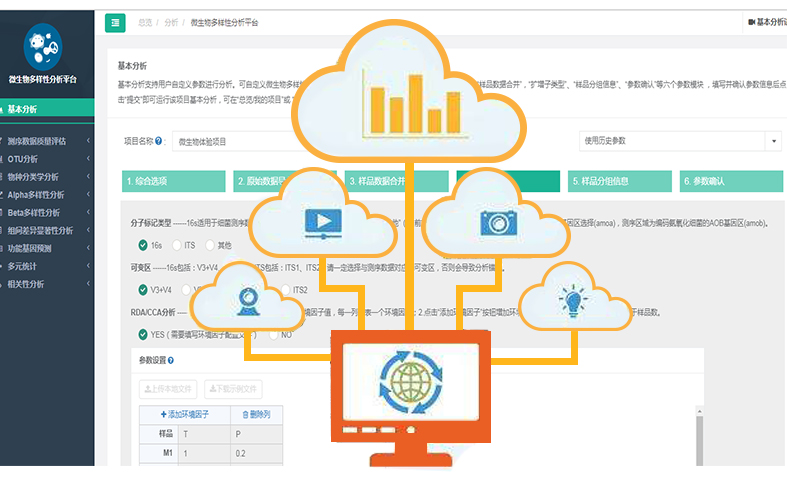
01月13 微生物分析平台个性化分析更新
功能预测分析 基于样品中的物种组成及丰度信息推测样品中表型类型和功能组成及差异。包含PICRUSt2功能预测、FAPROTAX功能预测、BugBase表型预测、Tax4Fun2功能预测和真菌FunGuild表型预测。 一. PICRUSt2功能预测 PICRUSt1软件依赖于Greengene数据库进行物种比对、功能数据输出。而Greengene数据库在2013年之后就停止了更新,距今为止已有7年。随着时间的推移,大量微生物基因组数据测序获得,而停止更新的Greengene数据库限制了PICRUSt的功能预测范围。对于近年来测序获得微生物功能功能信息无法进行预测,满足不了当前的研究需求,为了补上这块满足科研工作者的需求,PICRUSt团队于近期升级了软件,正式公布了PICRUSt2。 1)将待预测的OTU代表序列置于软件中已有的系统发育树中,而不是直接对OTU序列进行分类学注释; 2)不再基于GreenGene 16S数据库进行功能预测,其用于预测的参考基因组数据库相比先前也已扩大了10倍以上 参考文献:Douglas G M, Maffei V J, Zaneveld J, et al. PICRUSt2: An improved and extensible approach for metagenome inference. bioRxiv, 2019. 功能组成分析:统计各样品在不同分类层级上的功能组成。 注:横坐标为样品名称;纵坐标为功能相对丰度百分比。 功能差异分析:统计各样品或者各组在不同分类层级上的功能差异。 注:图中不同颜色代表不同的样品或分组。左图所示为不同功能在两个样品或者两组样品中的丰度比例,中间所示为95%置信度区间内功能丰度的差异比例,最右边的值为校正后p值。 二. FAPROTAX功能预测 FAPROTAX较适用于对环境样本的生物地球化学循环过程(特别是碳、氢、氮、磷、硫等元素循环)进行功能注释预测。FAPROTAX是根据已发表的文献手动构建的数据库,它把原核微生物的分类和代谢等功能对应起来,目前收集自4600多个原核微生物的80多个功能分组7600多条功能注释信息。 参考文献:Louca S, Parfrey L W, Doebeli M. Decoupling function and taxonomy in the global ocean microbiome[J]. Science, 2016, 353(6305): 1272-1277. 三. BugBase表型预测 BugBase是一种预测复杂微生物组内功能途径的生物水平覆盖以及生物可解释表型的方法。BugBase首先通过预测的16S拷贝数对OTU进行归一化,然后使用提供的预先计算的文件预测微生物表型。包括以下七方面:革兰氏阳性 (Gram...