
06月28 利用百迈客云挖掘海量公共测序数据的价值
近年来,随着测序技术的不断升级,测序通量越来越高,同时测序成本直线下降,高通量测序技术逐渐应用在科研、医疗、健康等各个研究领域。伴随着大数据处理关键技术的突破和数据共享等契机的发展,大数据将在促进生物学发展中发挥重要的作用。
测序技术在生物学研究中的发展分成三个阶段:
第一个阶段:Genome Center唱独角戏。在这个方向刚兴起的初期,人才储备较少,产出数据的成本也很大,只有少量的国家支持的Genome Center可以从事这方面的系统研究,做生物信息开发的目的也比较明确,就是为了完成基因组中心所承担的一些重大国家科研课题。
第二个阶段:高通量测序技术的进步和基因科技服务公司的崛起。当很多科学家看到了有那么多空白的山头待开发,同时数据产出的成本也大幅降低情况下,科学家们对组学研究都跃跃欲试,科技服务的市场需求就出现了,同时经过多年的人才储备,也有了一定数量的技术人员,为科技服务公司的出现打下了人力基础。这个阶段生物信息开发注重的是“pipeline”,这样能提高服务的效率,让公司能更好的发展。
第三个阶段:大数据的积累和丰富多样的个性化分析需求。随着NGS技术的不断发展,测序成本不断降低,数据积累也越来越快,同时随着科学家对数据理解的深入,科学家的思路大大开拓,各种个性化的数据分析想法大量涌入脑海,“personalized”成为了这个阶段的生物信息开发要满足的关键需求。目前,测序技术在生物学研究中的应用已经逐渐进入了第三个阶段,基因大数据的价值已经在很多方面超越了自己新测序项目的产出,同时传统基因科技服务公司提供的“pipeline”的分析结果已越来越难满足科研工作者的需求,一方面是分析工具和个性化的分析需求得不到满足;另一方面,孤立的单个项目的数据分析形式越来越不能满足科研的需要。能不能利用可视化的分析工具进行基因大数据的深入挖掘成为了能否在基因研究2.0时代成为佼佼者的关键因素。
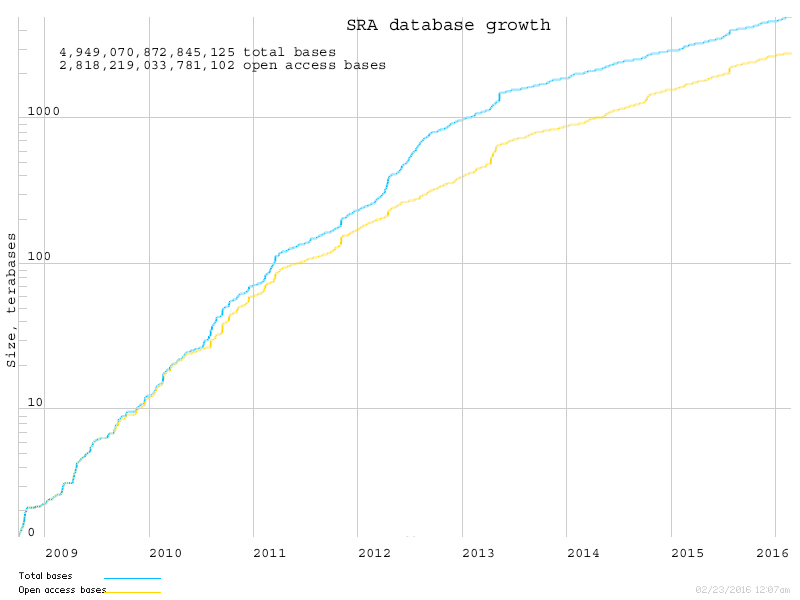
测序数据量呈指数级增长
Nature Reviews Genetics报道称,目前已发表的高通量测序数据利用率不足20%,很多有价值的信息被研究者所忽略,基于公共数据挖掘和分析将成为科学研究发展的趋势。例如,2015年 Nature Genetics发表的文章通过分析正常组织和各种常见类型癌症组织7256个转录组测序样本,43T转录组数据中鉴定得到58000个lncRNA;发现上千种lncRNAs 在癌症组织中特异表达,可以作为新的肿瘤标记物。2014年 Genome Biology 发表的文章通过研究13种组织和30个玉米转录组数据中LncRNAs的表达,鉴定出了20163个推测的LncRNAs, 并对LncRNAs的组织特异性的表达调控网络进行了解析。最有代表性的是最大的癌症基因信息的数据库,Cancer Genome Atlas/TCGA蕴藏着难以想象的宝贵信息,围绕TCGA已经有很多重量级文章出现,PubMed收录的利用其进行数据挖掘发表的文章已经超过1500篇,而且对它的使用必然愈发重要。可以说,已经公开的高通量测序数据如同“金矿”一般,具有极高的学术价值。然而,科研工作者一方面不方便获取符合自己需求的数据,数据的整合、分类、下载也会耗费较多的精力;另一个方面,缺少一个高效、易用的数据分析平台,自主搭建一个大数据分析平台,无论是硬件、软件,还是人力、财力都是巨大的挑战。这两个因素导致大量的基因数据沉睡在哪里,不能很好的挖掘出其中的价值。
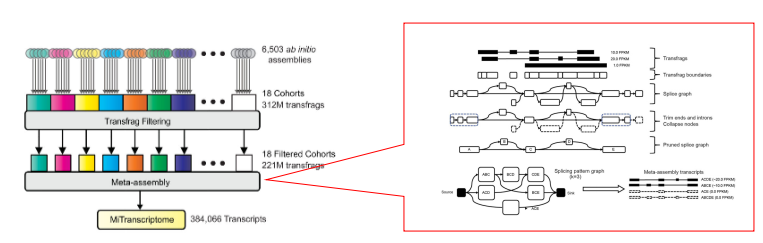
图1. 整合6503个来源18个器官的人类RNA-seq公共数据集,分开组装->混合组装(Meta-assembly)->merge后得到384,066个高可信度人类转录本,用于后续功能分析。
Iyer MK et al. The landscape of long noncoding RNAs in the human transcriptome. Nat Genet. 2015
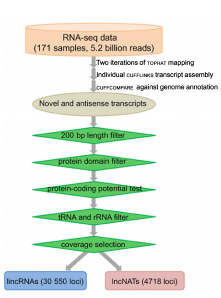
图2. 整合171个棉属RNA-seq公共数据集,基于海岛棉基因组进行棉属转录本重构,基于重构的转录本序列信息,从中鉴定得到35,268个lncRNA,用于后续棉纤维发育相关lncRNA鉴定。
Wang M et al. Long noncoding RNAs and their proposed functions in fibre development of cotton (Gossypium spp.). New Phytol. 2015
2016年3月,作为BMKCloud公共数据库项目的一部分,涵盖动物、植物、微生物的高通量测序数据库正式上线。百迈客云的合作伙伴现在可以在BMKCloud上免费访问该数据库了。在BMKCloud上将数10PB规模的高通量测序数据作为共享资源,用户无需下载数据,直接在BMKCloud上利用分析平台进行分析,加速了利用公共数据进行研究和探索的步伐。BMKCloud公共数据库项目的目标是建立植物、动物、细胞系、微生物、宏基因组、疾病高通量测序数据库以及肿瘤高通量测序数据库。目前,动物、植物、微生物高通量测序数据库正式上线。每个数据库中都收集和整理了全世界公开发表的各个物种高通量测序的原始数据及相关论文,为开展各个领域的研究提供准确、全面、最新的测序原始数据和相应文献的整合检索平台,并与各项可视化分析流程无缝对接。BMKCloud公共数据库项目的数据来源是NCBI的SRA、GEO等高通量测序的公共数据,数据来源可靠,涵盖重测序、转录组等各个研究领域(以水稻为例,目前已收录的基因组数据集29910个,转录组数据集2100个)。

图3. BMKCloud公共数据库中常见研究物种数据收录
BMKCloud公共数据库项目建立的目的也是让科研工作者更高效、更快速的利用公共数据,提高公共数据的利用效率。使大数据的挖掘不仅仅局限于癌症的研究,而能够在动物、植物、微生物等研究中都得到很好的利用。科研工作者可以根据物种、类型、研究性状、研究结果、测序方法等设置快速检索感兴趣的数据,所有数据均可一键导入平台中“我的数据”中保存。然后利用百迈客云提供的20大数据分析平台和100多款工具进行数据的可视化深度挖掘,高效、快速的挖掘基因大数据。
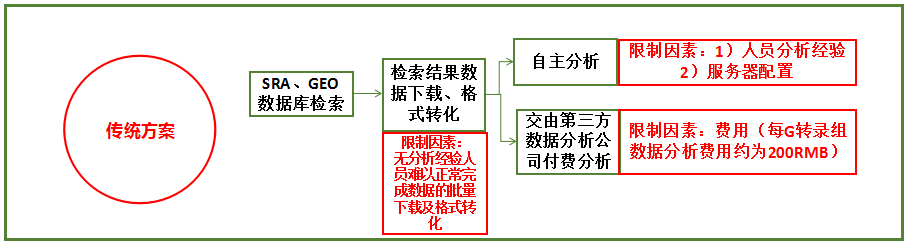
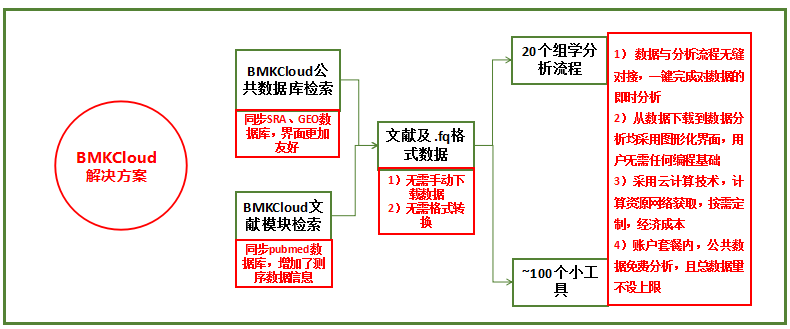
图4. 公共数据分析传统方案 vs BMKCloud解决方案
百迈客云(BMKCloud)是一个基于基因大数据的挖掘,管理和科研协作的整合平台。可以使不具备生物信息分析基础的用户,快速掌握独立的生物信息分析能力,实现高通量数据的深度解读。百迈客云提供:20大生物信息分析平台(重测序、外显子、转录组、非编码RNA等),基本分析+个性化分析,轻松实现原始数据到生物学问题准确解读的转化;100多款数据分析工具,涵盖数据处理、文件操作、序列比对、基因注释、进化分析和图表制作等,满足个性化数据分析需求。百迈客生物云平台,用户定位是非生物信息背景的生物学家,从2014年5月开始开放试用到2015年10月份正式商用。对于广大的科研用户而言,百迈客生物云是一个完整的交钥匙(Turn-Key)解决方案,用户只需要开通云平台账号,就拥有了属于自己的生物信息分析平台,百迈客云已经成为“基因科技服务2.0时代”(基因大数据可视化即时自主分析的时代)科研工作者受欢迎的生物云平台。
附1公共数据整合分析经典思路总结部分文献
发育调控研究中,利用公共数据完善取样时间点或取样部位
- YU C et al. Transcriptome dynamics of developing maize leaves and genomewide prediction of cis elements and their cognate transcription factors. Proc Natl Acad Sci. 2015
- Patil G. et al. Soybean (Glycine max) SWEET gene family: insights through comparative genomics, transcriptome profiling and whole genome re-sequence analysis. BMC Genomics.2015
比较转录组分析中,补充其他物种、组织部位、发育时间点等相关数据
- Fu Y et al. Dissection of early transcriptional responses to water stress in Arundo donax L. by unigene-based RNA-seq. Biotechnol Biofuels. 2016
- Lynch VJ et al. Ancient transposable elements transformed the uterine regulatory landscape and transcriptome during the evolution of mammalian pregnancy. Cell Rep. 2015
- Analysis of Bos taurus and Sus scrofa X and Y chromosome transcriptome highlights reproductive driver genes. Oncotarget. 2017
ncRNR-mRNA联合分析中,补充mRNA或者ncRNA数据
- Xu W et al. Genomic DNA Methylation Analyses Reveal the Distinct Profiles in Castor Bean Seeds with Persistent Endosperms. Plant Physiol. 2016
- Liu X et al. MicroRNA-mRNA regulatory networking fine-tunes the porcine muscle fiber type, muscular mitochondrial respiratory and metabolic enzyme activities. BMC Genomics. 2016
转录本构建项目中,整合大量公共数据,构建较为全面的物种转录本序列文库
- Wang M et al. Long noncoding RNAs and their proposed functions in fibre development of cotton (Gossypium spp.). New Phytol. 2015
- Iyer MK et al. The landscape of long noncoding RNAs in the human transcriptome. Nat Genet. 2015


