
03月02 水稻信息网(RIGW):一个全面的籼稻基因组生物信息学平台
英文名:Rice Information GateWay (RIGW): a comprehensive bioinformatics platform for indica rice genomes
中文名:水稻信息网(RIGW):一个全面的籼稻基因组生物信息学平台
发表杂志:MOLECULAR PLANT
影响因子:8.827

Oryza sativa subsp.籼稻和O. sativa subsp. 粳稻是亚洲栽培稻的两个亚种,其中籼稻的种植面积更广,遗传多样性也更高。在过去的几年,水稻注释项目数据库(RAP-DB)(Ohyanagi等,2006)和密歇根州立大学水稻基因组注释项目(MSU-RGAP)(Ouyang等,2007)是两个受欢迎的数据库, 基于粳稻品种Nipponbare(国际水稻基因组测序项目,2005)的统一参考基因组的基因组和转录组数据。北京基因组研究所的水稻信息系统(BGI-RIS)(Zhao等,2004)是籼稻栽培品种93-11的可用资源,但由于缺乏高质量的籼稻参考基因组,其应用受到限制。 为了弥补这一缺点,研究人员构建了一个综合全面的平台:Rice Information GateWay(RIGW,http://rice.hzau.edu.cn/),提供基因组学,转录组学,蛋白质 – 蛋白质相互作用(PPIs),代谢网络,代谢物和计算工具,以最新的籼稻珍山97(ZS97)和明恢63(MH63)(Zhang et al.,2016)为参考基因组。RIGW通过直观的Web的界面,为水稻研究提供丰富的基因组学和其他组学数据。
RIGW在Linux操作系统和Apache Tomcat Web服务器(http://tomcat.apache.org/)中实现。 所有的基因组数据,注释,同系物,基因表达,PPIs,代谢物和文献存储在MySQL数据库(http://www.mysql.com/)中。 图1A显示了RIGW中的体系结构,一些有代表性的资源和计算工具。
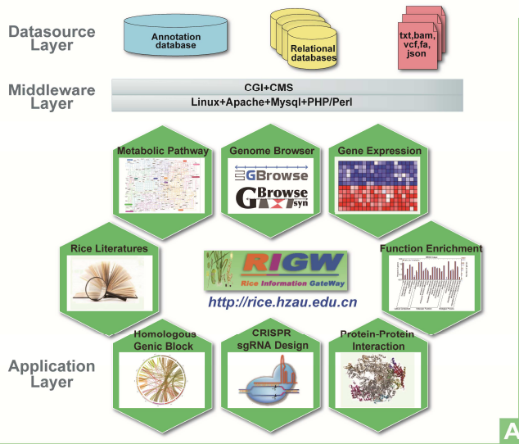
图A RIGW体系结构
RIGW上部署GBrowse(https://github.com/GMOD/GBrowse)用于ZS97和MH63基因组和转录组数据的可视化(图1B),分别选择了包括基因注释和标记叶子、圆锥花序以及芽中的RNA-sequencing。
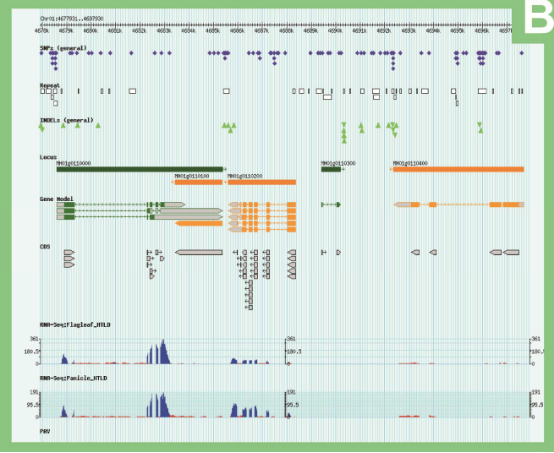
图B GBrowse
另外,为ZS97,MH63和Nipponbare基因组之间的比较分析提供了Gbrowse_synteny工具(图1C),所有位于同一染色体区域的相应注释可以很容易的并行显示出来(每个基因组都可以设置为参考)。
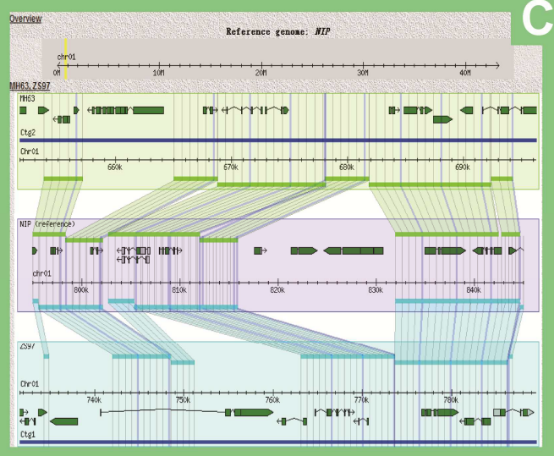
图C Gbrowse_synteny工具
研究人员开发了一个灵活的查询界面来高效地检索和图形化显示各种数据。 例如,提供关键词的搜索引擎,用户输入关键词(例如基因位点,基因功能)就可以链接到详细页面(例如基因位置,基因结构,可变剪接,其他品种水稻中的同源物, 核苷酸和氨基酸序列,基因表达水平等)。 此外, Gene Ontology(Harris等,2004),InterPro域信息,预测亚细胞定位和蛋白质 – 蛋白质相互作用以及外部数据库链接在可以获得的情况下都列在搜索结果中(图1D)。 提供局部序列比对搜索工具(BLAST)作为碱基序列的搜索引擎,可以查找在籼稻ZS97,MH63,93-11和粳稻日本晴中的同源序列,比对结果通过图形和文本的格式呈现。
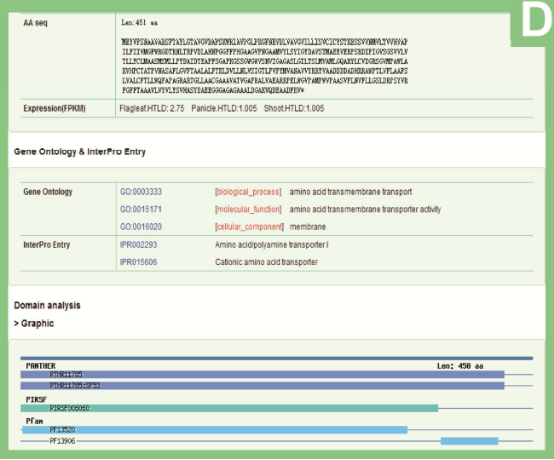
图D 搜索结果展示
在补充表1和RIGW主页中列出了ZS97和MH63基因组特征。研究人员根据相关文献,在不同品种水稻中手动收集了2000多个克隆的基因,和2500多个具有详细注释信息的水稻代谢物。利用CREP(http://crep.ncpgr.cn/)的数据,研究人员建立了一个友好的网络界面,用于查询和显示ZS97,MH63及其杂种汕优63(SY63)生命周期中39个组织的基因表达水平。对于给定的基因,可获得的所有组织表达量信息,这极大地促进了其表达模式的研究。由于全基因组PPI网络对研究整体细胞反应非常有用,研究人员从公共数据库收集了1,871,563个非冗余水稻PPIs(其中929个为实验确定的PPIs),包括PRIN(Gu et al.,2011), RiceNet(Lee等,2015)以及RIGW中的相关文献。用户可以在PPI搜索页面上提交一个或多个ZS97 / MH63 / Nipponbare的基因ID,查询相互作用的蛋白质,可以帮助揭示不同种类不同功能蛋白质之间的关系。查询蛋白及其互作用Cytoscape(http://www.cytoscape.org/)可视化软件,不同颜色的点表示不同途径分类(图1E)。此外,所有日本晴,ZS97和MH63的PPIs都可以从“下载”模块下载。

图E Cytoscape可视化显示蛋白互作网络图
KEGG代谢通路图是表示代谢反应网络信息的图表,每张图都汇总了已发表文献中的实验结果(Kanehisa等,2012)。 基于KEGG Orthology(KO)组,研究人员获得了ZS97和MH63基因组中的KEGG同源序列,以及它们的代谢途径。 ZS97和MH63的代谢通路包括四个类别(代谢,遗传信息处理,环境信息处理和细胞过程),每个类别包含许多途径。 当选择特定的途径时,在ZS97和MH63中KEGG同源序列的酶/蛋白质用绿色标出(图1F)。
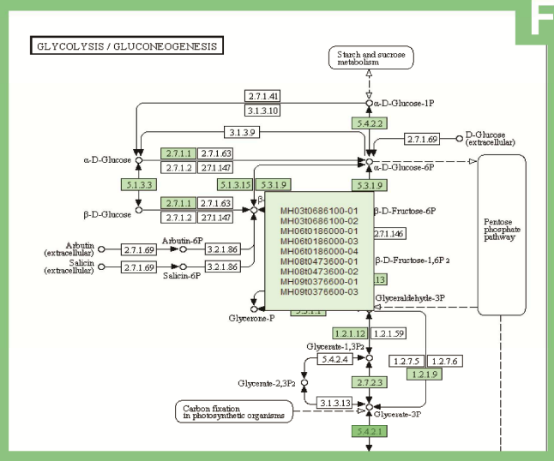
图F KEGG代谢通路图
在RIGW中集成了一系列计算工具,用于比较水稻和其他植物的进化、功能分析。 OrthoMCL(Li等人,2003)被用于鉴定植物基因组中的同源物,包括拟南芥,短柄草,玉米,葡萄和高粱。研究人员可以通过OrthoMCL(e值:1e-5)来鉴定假定的同源序列和相应的逻辑关系,并获得水稻和上述植物中产生紧密相关蛋白质的不连续簇,共鉴定了48,515个假定的直系同源组,并保存在RIGW中,可以从“下载”模块获得。研究人员通过MCscanX(Wang et al.,2012)确定了在染色体中的同源基因对(e值<1e-10),以及同源基因座以显示ZS97和MH63基因组中的部分复制区域(图1G)。研究人员还提供了基因ID转换工具来转换ZS97,MH63,93-11和日本晴之间的直系同源基因ID。此外,该平台还提供KEGG / GO富集,GO分类工具,可进行功能富集分析。
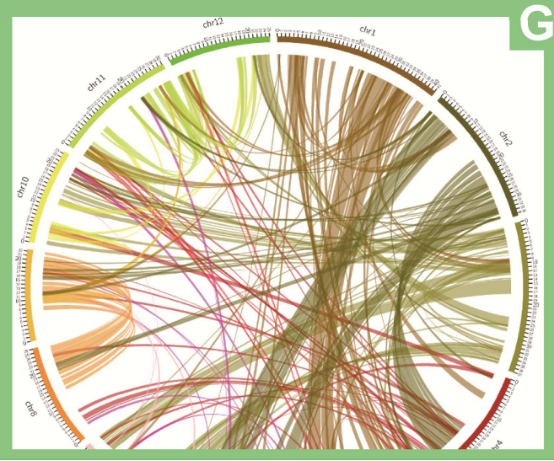
图G OrthoMCL植物基因组中的同源物鉴定
为了方便不同水稻品种的基因编辑,研究人员整合了CRISPR-P 2.0(Liu et al.,2017)用于设计各种规律成簇的间隔短回文重复(CRISPR)—Cas系统的指导性RNA序列,实验结果如图1H所示。
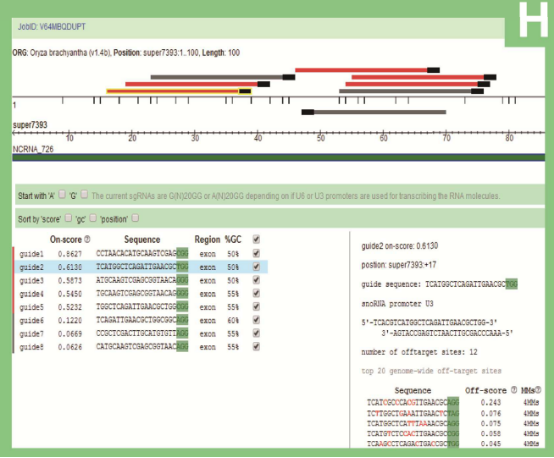
图H CRISPR-P 2.0基因编辑工具
最后,RIGW还提供了一个文本挖掘工具,用户可通过基因名称或关键词搜索获得的27,831个水稻相关文献,文献来源于PubMed(图1I)。
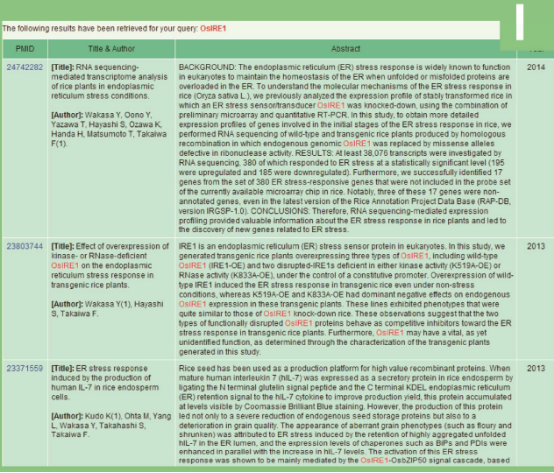
图I 文本挖掘工具
总而言之,研究人员建立了一个全面的生物信息学平台RIGW,提供可在GBrowse视图下查看的ZS97和MH63基因组以及其他组学数据。 RIGW还提供了籼稻,粳稻和其他植物的同源染色体。 并且为用户提供了友好的网页界面来显示水稻中预测的PPIs,ZS97 / MH63的代谢途径,CRISPR-Cas单引导RNA设计工具,和GO富集。 此外,所有的基因组序列和注释都可以自由访问,同时,还提供与其他公共数据库的有效链接。研究人员即将整合更多的可用资源,并通过新的工具扩展其功能,使RIGW成为一个综合的生物信息学平台,为水稻研究人员服务。 RIGW免费使用网址是http://rice.hzau.edu.cn/。
百迈客数据库搭建业务
构树数据库(papyrifera.biocloud.net)
大豆数据库(soybean-resources.cn)
草业数据库(grassgene.biocloud.net)
参考文献:
Song J.-M., Lei Y., Shu C.-C., Ding Y., Xing F., Liu H., Wang J., Xie W.,Zhang J., and Chen L.-L. (2017). Rice Information GateWay (RIGW): A Comprehensive Bioinformatics Platform for Indica Rice Genomes. Mol. Plant. doi: 10.1016/j.molp.2017.10.003.
转录调控事业部 赖娟娟 | 文案
吴戈宇 | 审核
图片来自网络,侵删


