03月20 百迈客云平台——助您轻松搞定聚类热图绘制
聚类热图是以热图的形式来进行聚类结果的展示,可以直观的从图上分析哪些数据具有相似性,哪些数据差异较大。百迈客云(BMKCloud)免费推出的聚类热图小工具主要针对矩阵文件(如不同样品的基因表达量、样本相关系数矩阵等)进行聚类分析及图片绘制,并且可以根据研究情况对绘图结果进行交互式操作(如筛选数据,调整图片配色等)。
应用场景:
使用矩阵数据文件进行热图绘制,通常可以对矩阵数据进行筛选,归一化和聚类等处理,多用于不同样品间基因表达水平聚类分析。主要应用在真核有参转录组、真核无参转录组、微生物多样性等数据分析中。
操作步骤:
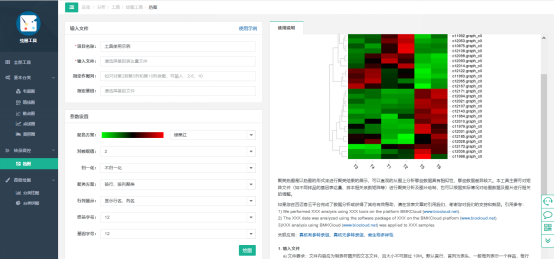
登录百迈客云首页(www.biocloud.net)——分析——工具——绘图工具——热图

操作方法:
1. 输入文件
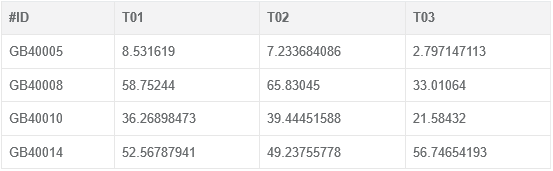
(1)文件要求:文件内容应为制表符隔开的文本文件,且大小不可超过 10M。默认首行、首列为表头,一般每列表示一个样品,每行表示一个基因,也可统计其他含义的数据矩阵。除表头外,参与统计绘图的内容应为纯数字,文件范例如下:
(2) 指定作图列:可对指定列绘图,如只对第 2 到第 5 列和第 10 列作图,可输入:“2-5,10”。若想按特定顺序绘图,需用逗号将绘图列按序列出,并在下方参数中取消按列聚类,如“6,3,2,5,4”。
(3) 指定基因:可输入基因列表文件,系统会自动过滤空行或以#开头的行并提取第一列作为指定基因,结合上方输入的矩阵文件进行统计绘图。
2. 参数设置
(1)配色方案:设置绘图所采用的配色。可以选择预制方案,也可以根据实际实际需要自定义配色方案。
(2) 对数取值:对文件数据取对数后再绘图。取对数可以有效解决数据取值范围过大导致的配色问题。
(3) 归一化:对行或列进行归一化处理。可最大程度地呈现每行或每列的变化信息,避免超高值掩盖其他数据的变化。绘制基因表达量热图时,常按基因归一化。
(4) 聚类方案:可选择是否按行、列聚类。若按特定样本顺序绘制热图,可取消按列聚类。
a行列显示:可选择是否显示行、列 ID。
b 样品、基因字号:可调节行、列 ID 的字体大小。
3 注意事项
(1) 聚类分析涉及运算分析,当分析的基因数或样品数较多时,绘图时间可能较长,请待任务完成后点击预览查看、调整图片。
(2)如果选择对数据取对数,会自动将取值为 0 的数据转化为接近于 0 的小数。
4. 结果展示
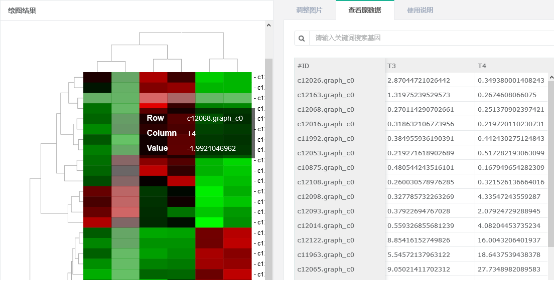
(1) 绘图结果:绘图结果展示区为您展示初始或调整后的绘图结果,同时,可通过图片区域的交互操作查看对应的数据信息,如:
a 鼠标悬停于图中相应数据格,可显示其对应的行名、列名和数值。
b 点击图中相应的行名或列名,在“查看原数据”页会高亮显示对应行或列。
c 选中行聚类树某分枝后,可在“查看原数据”也筛选出对应行并下载。
(2)全屏预览:点击图片右上角的缩放按钮,可全屏预览绘图结果。选中聚类树某分枝后点击缩放按钮,可在全屏预览时高亮对应的分枝,以便尽快找到目标区域。
(3)图片下载:点击预览区右上角的“下载”按钮,可保存 SVG 或 PNG 格式的绘图结果
(4)调整图片
a 配色方案:设置绘图所采用的配色。可以选择预制方案,也可以根据实际实际需要自定义配色方案。
b 对数取值:对文件数据取对数后再绘图。取对数可以有效解决数据取值范围过大导致的配色问题。
c 归一化:对行或列进行归一化处理。可最大程度地呈现每行或每列的变化信息,避免超高值掩盖其他数据的变化。绘制基因表达量热图时,常按基因归一化。
d 聚类方案:可选择是否按行、列聚类。若按特定样本顺序绘制热图,可取消按列聚类。
e 行列显示:可选择是否显示行、列 ID。
f 样品、基因字号:可调节行、列 ID 的字体大小。
(5)查看数据
a聚类数据:显示聚类后数据矩阵的前100行,样品和基因顺序均与左侧图中一致。
b 搜索基因:可在搜索栏中输入关键词,搜索相应基因或基因集。
5.案例展示
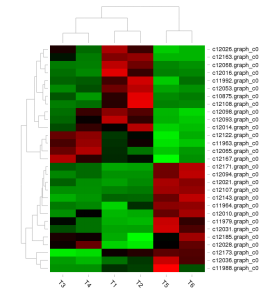
2017年,中国水产科学院喻达辉老师在百迈客云平台上对合浦珠母贝免疫相关数据进行分析,2篇文章先后分别发表于同一个杂志《Fish & Shellfish Immunology》,这两篇文章中都进行了聚类热图分析。第一篇文章中研究人员将免疫相关差异表达基因按照不同时期进行分层聚类分析,第二篇文章中研究人员将不同时期的unigene进行了聚类热图分析。为后续的研究结果的呈现做了较好的铺垫作用。
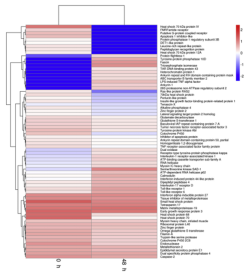 免疫相关差异表达基因按照不同时期进行分层聚类
免疫相关差异表达基因按照不同时期进行分层聚类
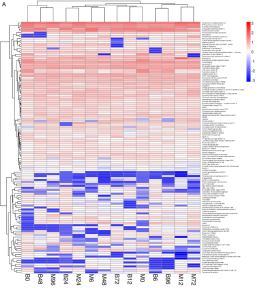
不同时期unigene聚类热图
百迈客云平台是由北京百迈客生物科技有限公司开发,集生物信息分析软件、数据库以及云计算为一体的生物大数据分析平台。功能强大,操作简单的聚类热图就介绍到这里了,后期云平台会为大家准备更多实用小工具的操作指引,如果您在操作过程中遇到任何问题都可以联系咱们的云客服,欢迎点击屏幕右下方客服图像进入咨询环节。
参考文献
1.Wei J, Liu B, Fan S, et al. Differentially expressed immune-related genes in hemocytes of the pearl oyster Pinctada fucata, against allograft identified by transcriptome analysis[J]. Fish & Shellfish Immunology, 2017, 62:247-256.
2.Wei J, Fan S, Liu B, et al. Transcriptome analysis of the immune reaction of the pearl oyster Pinctada fucata to xenograft from Pinctada maxima.[J]. Fish & Shellfish Immunology, 2017.


